A pesar de que todas las células del cuerpo albergan los mismos genes, en cualquier célula, algunos genes son silenciosos y otros son expresivos (es decir, el código genético es convertido en productos génicos – ARN y proteínas). Una mezcla particular de productos de expresión génica da a las células una identidad individual. Uno de los retos más difíciles en la investigación biomédica moderna es revelar los planos moleculares de células individuales. Tecnologías actuales sólo permiten la evaluación de la expresión génica promedio de millones de células, y desafortunadamente, promediar la expresión de un grupo grande de células puede ocultar las diferencias en expresión génica entre células individuales.
Para tratar este asunto, un equipo de investigación de la Universidad Estatal de Arizona dirigido por Stuart Lindsay, Profesor de Física y Química y Bioquímica; Hao Yan, Profesor de Química y Bioquímica; y asistidos por Yung Chang, Profesor Asociado de Inmunología, diseñaron un nanodispositivo de ADN autoensamblable para medir los productos de expresión génica en pequeñas muestras.
Construcción de Nanoestructuras de ADN
El ADN, llamado con frecuencia el bloque constructor de la vida, está siendo explorado cada vez más como un material para construir nanoestructuras artificiales. Desde una perspectiva ingenieril, el ADN es un material deseable, gracias a la combinación única de estabilidad química y física, rigidez, y flexibilidad. Además, el ADN puede ser sintetizado rápida y económicamente, y sus dimensiones son ideales para la construcción de estructuras a pequeña escala.
En una doble hélice de ADN, dos hebras individuales se sostienen juntas a través de enlaces entre componentes del eje del ADN llamados bases (p. ej., la base A se une a la base T, y la base C a la base G). Este principio de emparejar bases se utiliza en la técnica “origami ADN”, donde numerosas hebras cortas individuales de ADN actúan como grapas para doblar una hebra larga individual de ADN llamada andamio. Al programar la secuencia de las grapas para que se unan en sitios específicos, el andamio se puede doblar prácticamente en cualquier forma.
El equipo de Lindsay y Yan creó una teja rectangular formada de un ADN viral y más de 200 hebras ayudantes cortas. Algunas de estas hebras cortas llevaban un saliente o sonda de ADN de hebra individual. Cada sonda contenía una secuencia única que actuaba como un pegamento molecular para capturar ARN mensajero (mARN), el químico mensajero entre los genes y proteínas. Cuando la sonda capturó el mARN, se formó una hélice rígida doble de ADN-ARN en forma de V. El cambio local en rigidez fue fácilmente detectado por un microscopio de fuerza atómica que tomaba imágenes de la superficie de la teja. Esta teja básica puede ser modificada para obtener un perfil completo de expresión génica, mediante la detección de muchos genes simultáneamente. “Es un proceso de ensamblaje de una-olla. Si utilizas una secuencia más grande de ADN viral o una secuencia más grande de ADN de hebra individual, puedes hacer una teja más grande. También puedes hacer tejas individuales en tubos separados y luego combinarlas para hacer una super-teja que contenga más sondas accesibles”, explica Yan.
Tejas de ADN contra Microarreglos
Una teja de ADN con arreglos de sondas sirve como superficie de unión y como reactivo químico simultáneamente. Debido a que las reacciones se manifiestan en solución, se evitan los obstáculos de unir biomoléculas a superficies sólidas, algo común en la tecnología de microarreglo. “Comparado con el sistema de microarreglo, donde conoces la densidad promedio del arreglo pero no conoces realmente el espacio exacto entre las sondas cercanas, aquí puedes controlar la geometría y organizar las sondas de manera específica. Debido a que este arreglo es soluble en agua, se puede reducir a volúmenes pequeños. La meta principal sería hacer esto compatible con el volumen de una célula individual. Es difícil reducir el microarreglo a ese nivel”, comenta Yan.
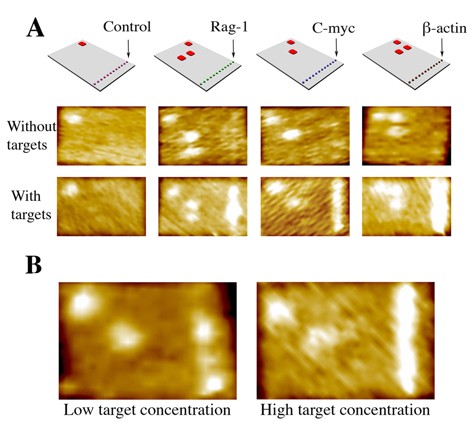
Se puede hacer un perfil del mARN de células individuales ya que aún las cantidades más pequeñas de ácidos nucleicos se pueden amplificar en un tubo de ensayo para obtener suficiente material para análisis. Por el contrario, las proteínas no pueden ser amplificadas fuera de la célula viva, y, de acuerdo con Lindsay, “estamos estancados con ver, en el mejor de los casos, a cientos de miles de células utilizando espectroscopía masiva. Por el contrario”, dice él, “yo veo esta tecnología [teja de ADN] como la que abre la posibilidad de hacer mediciones de arreglos a nivel de célula individual. Probablemente existe una gran variación célula-a-célula en la expresión de las proteínas con un número bajo de copia. Al observar a los individuos de una población en lugar de el promedio de la población, quizá obtengas mejores respuestas a preguntas como, ‘¿Por qué fue que la célula X se hizo tumorigénica en una población de células todas sujetas al mismo estrés ambiental?’”.
Aplicaciones Potenciales de las Tejas de ADN
Lindsay advierte que, tal como con cualquier tecnología dramáticamente nueva, habrá obstáculos que superar antes de que encuentre aplicación en la clínica, pero las posibilidades son extraordinarias. Los laboratorios de Lindsay y Yan están desbordando de ideas.
Usando aptámeros – moléculas sintéticas diseñadas para capturar objetivos moleculares con muy alta selectividad – adheridos a una teja de ADN, se pueden capturar proteínas que contienen varios grupos químicos (modificaciones) en su superficie. Estas modificaciones juegan un papel primordial en el señalamiento celular, pero son difíciles de detectar con técnicas existentes, especialmente en muestras pequeñas. “Podemos reconocer con gran especificidad un cambio muy pequeño en un residuo aminoácido en una proteína muy grande. Al combinar la experiencia química con la experiencia de autoensamblaje, creemos que podemos hacer algunos productos de diagnóstico nuevos muy radicales”, añade Lindsay.
Además, se pueden usar andamios de ADN y aptámeros para crear organelos artificiales (es decir, subunidades celulares especializadas, como la mitocondria – la central energética de la célula). El construir sistemas biológicos simples permitirá a los investigadores ganar una mejor comprensión de sus trabajos internos.
Otro proyecto involucra el hacer una máquina muy sofisticada a nanoescala que hace más de lo que hacen los medicamentos simples. Se podrían utilizar nanocanales artificiales con sondas múltiples para conectar células inmunes con células cancerosas y mejorar su habilidad para matar tumores.
Los doctores Yan, Lindsay, y Chang están actualmente persiguiendo estas metas. “Esta tecnología ofrece una nueva manera de pensar. Todos los sistemas vivos son autoensamblables y solubles en agua. ¿Por qué no sólo utilizar el concepto de autoensamblaje y hacerlo compatible con lo que sea que ya existe en sistemas vivos?”, se cuestiona Yan. “Es casi nanotecnología de cocina; es muy simple. Aunque no estamos seguros de cuáles serán las primeras aplicaciones comerciales, habrá sin duda aplicaciones”, añade Lindsay.
Referencias
Lin C, Liu Y, Rinker S, Yan H. DNA tile based self-assembly: building complex nanoarchitectures. Chemphyschem. 2006 Aug 11;7(8):1641–7.
Chhabra R, Sharma J, Ke Y, Liu Y, Rinker S, Lindsay S, Yan H. Spatially addressable multiprotein nanoarrays templated by aptamer-tagged DNA nanoarchitectures. J Am Chem Soc. 2007 Aug 29;129(34):10304–5.
Ke Y, Lindsay S, Chang Y, Liu Y, Yan H. Self-assembled water-soluble nucleic acid probe tiles for label-free RNA hybridization assays. Science. 2008 Jan 11;319(5860):180–3.
Rinker S, Ke Y, Liu Y, Yan H. Self-assembled DNA nanostructures for distance- dependent multivalent ligand-protein binding. Nat Nanotechnol. 2008 Jul;3(7):418–22.
